Tävling i maskininlärning ska få fram bästa modellerna för klassificering av mänskliga proteiner
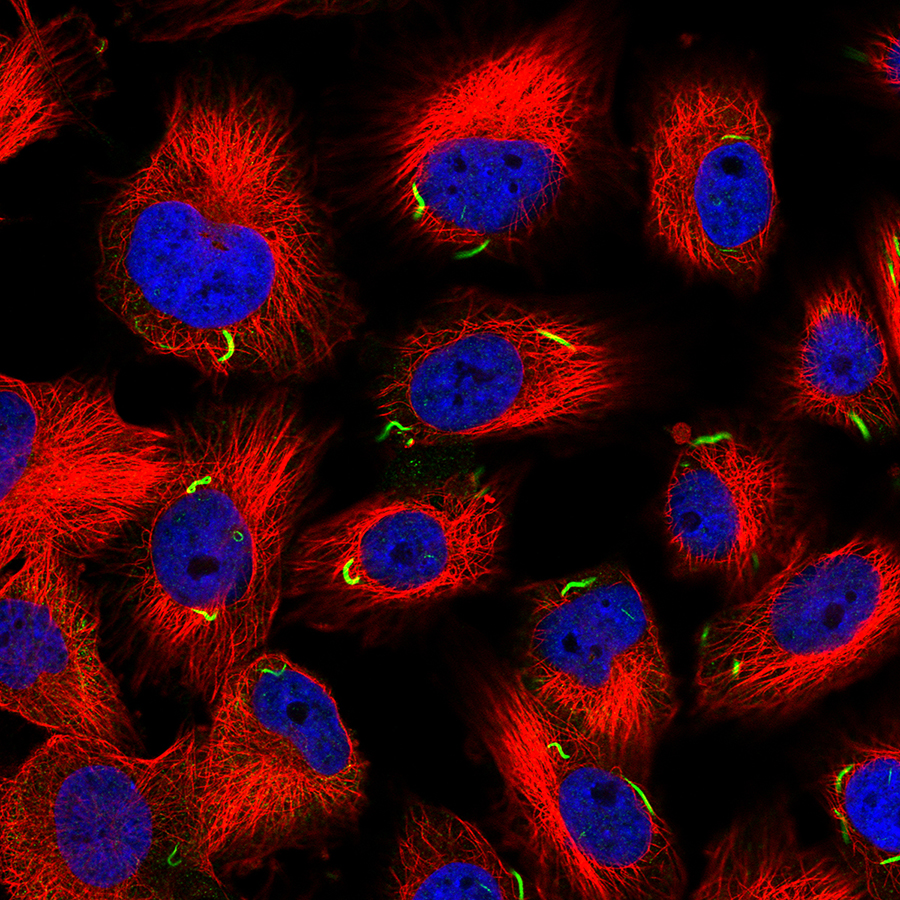
Forskare inom biomedicin på KTH har tillsammans med Kaggle – världens största community för forskare inom datavetenskap – lanserat en tävling inom maskininlärning. Priser om totalt 37 000 USD kommer att delas till dem som tar fram de bästa algoritmerna som klassificerar proteiner i bilder av människans celler. Tävlingen pågår under 90 dagar på Kaggles webbplats med start idag.
Bakom utmaningen står Human Protein Atlas, ett framstående svenskt initiativ inom bioteknik där fokus ligger på att kartlägga alla mänskliga proteiner i celler, vävnader och organ. Mikroskopbilder som visar var i våra celler proteiner finns används i hög utsträckning inom biomedicinsk forskning. Dessa bilder kan sitta på nyckeln till nästa genombrott inom medicin. Bilderna kan numera genereras snabbare än forskarna hinner analysera dem.
– Det finns ett stort behov av utökad automatisering inom biomedicinsk bildanalys för att förbättra förståelsen av människans celler och sjukdomar, säger Emma Lundberg , forskare på KTH som leder Cell Atlas-projektet – en del av Human Protein Atlas – vid forskningscentret Science For Life Laboratory (SciLife Lab).
Tävlingen ”The Human Protein Atlas Image Classification Challenge” bjuder in deltagare för att träna maskininlärningsmodeller i syfte att klassificera mönster för proteinuttryck i bilder av människoceller. De deltagare som tar fram de bästa algoritmerna får dela på 37 000 USD.
Proteiner är molekyler som utför en mängd olika funktioner i våra celler, konstaterar Emma Lundberg. Hennes studier har visat att hälften av alla människans proteiner finns på flera ställen i cellen. Något som går på tvärs mot den klassiska bilden att de flesta proteiner finns på ett ställe.
Historiskt sett har denna typ av bildklassificering av proteiner begränsats till enstaka mönster i en eller ett fåtal celltyper. Men för att verkligen förstå den mänskliga cellens komplexitet måste man ta fram modeller där man kan klassificera blandade mönster i många olika celltyper.
I en studie som genomfördes nyligen av Emma Lundberg och hennes Cell Atlas-team klassificerades dessa bilder genom både medborgarforskning och artificiell intelligens. Dock är dessa modeller ännu inte lika bra som de modeller experter tar fram (Sullivan et al, Nature Biotechnology, oktober 2018). Nu hoppas Emma Lundberg och hennes kollegor få hjälp av Kaggle-deltagare för att ta fram modeller som är lika bra eller bättre än experternas modeller.
Human Protein Atlas kommer att använda modellerna som tas fram genom Kaggle-tävlingen för att automatisera denna process direkt i deras mikroskopisystem som används för förbättrad analys av bilderna i Human Protein Atlas.
– Jag ser fram emot att se de olika innovativa sätt som man angriper problemet att klassificera dessa bilder med. Den här metoden har potential att förbättra studier av proteiner och hur celler fungerar och öka vår förmåga att förstå människans biologi och sjukdomar, säger Emma Lundberg.
Läs mer om tävlingen på Kaggle.
David Callahan/Håkan Soold
